Windows(32X & 64X)系统下的本地BLAST安装方法
Windows(32X)系统下单机版BLAST安装方法
1.下载安装文件:
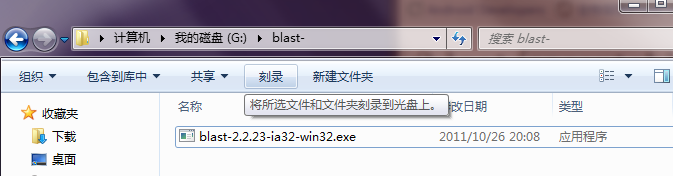
以blast-2.2.23-ia32-win32.exe为例,将此安装文件放至指定目录,以G:\blast-\为例,如图所示:
2.运行安装程序:
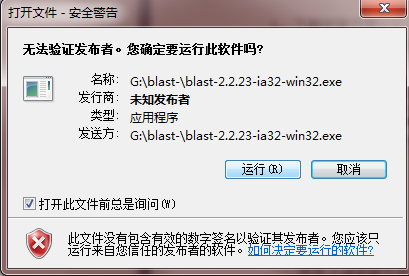
双击上述安装文件,单击运行:
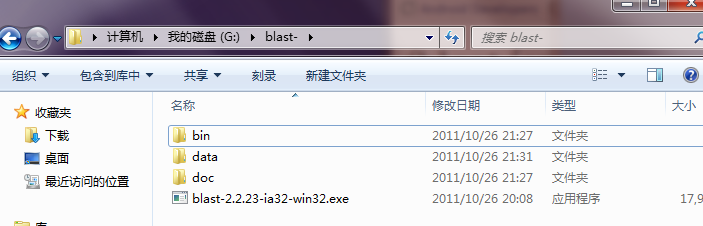
程序会自动在blast-文件夹下生成3个文件夹:\bin\、\data\和\doc\:
3.添加配置文件:
在桌面(任意可以新建文件的地方)新建一个.txt文件,然后将其重命名为NCBI.ini,在提示更改后缀名的对话框中点是。打开NCBI.ini,在其中写入如下两行内容:
[NCBI]
Data="path\data\"
上边的path是你的blast安装路径,在本例中为G:\blast-,因此,NCBI.ini中的内容为:
[NCBI]
Data="G:\blast-\data\"
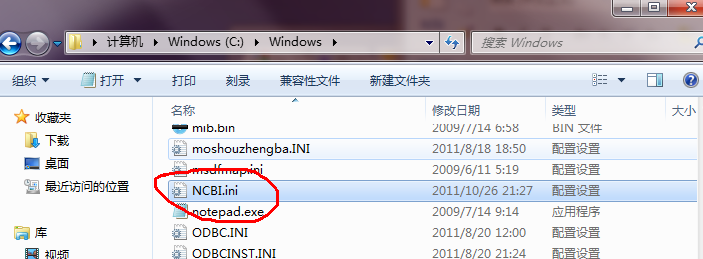
写完后保存,然后将该文件复制至C:\Windows目录下:
至此,本地blast-2.2.23-ia32-win32安装完毕。
4.导入数据库:
从ftp://ftp.ncbi.nlm.nih.gov/blast/db/上,可下载各类数据库文件,下载完毕后,将其解压至G:\blast-\data\目录下。
注意事项:
1.NCBI.ini中的路径为blast所在安装路径;
2.此安装办法适用于指定版本,对于blast+版本不适用,若想安装新版本,可自行到网站查阅安装办法;
附:运行示例:
1.打开cmd命令行;
2.通过cd命令到达安装目录的bin\目录下

3.通过dir命令查看全部可执行的子程序:
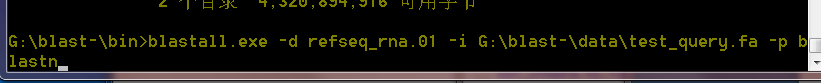
4.使用blastall.exe进行比对
输入blastall.exe -d refseq_rna.01 -i G:\blast-\data\test_query.fa -p blastn 该命令各部分的含义为:
- blastall.exe:blast主程序;
- -d refseq_rna.01:选择refseq_rna.01为被搜索的数据库,其数据已存至G:\blast-\data\下;
- -i G:\blast-\data\test_query.fa: 选择test_query.fa为查找序列文件,注意,查找文件应输入其绝对路径,但数据库文件不用。
- -p blastn:选择blastn为匹配方法,blastn为核酸-核酸匹配,想获取其他程序信息,可直接输入blastall.exe命令查看帮助信息。
程序部分运行结果为:
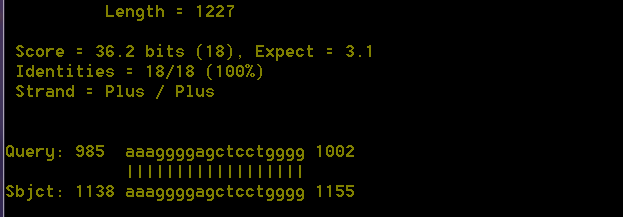
上述截图仅为一部分。
5.输入blastall.exe命令查看帮助信息:
Windows(64X)系统下单机版BLAST安装方法
1.blast的下载与安装
- 程序下载:访问blast本地软件包链接 blast_latest 下载适合自己系统的blast版本,这里我选择 ncbi-blast-2.2.28+-win64.exe。
- 安装流程:下载完毕后,双击安装到C:\Blast,生成bin和doc两个子目录,其中bin是程序目录, doc是文档目录,这样就安装完成。
- 用户环境变量设置:右键点击“我的电脑”-属性,然后“高级系统设置”选项-“环境变量”,在用户变量下方点击“新建”-变量名:BLASTDB,变量值:C:\Blast\db(即数据库路径)。在系统变量下方“Path”添加变量值:C:\Blast\bin。
- 查看程序版本信息:点击window的"开始"菜单,在运行中输入cmd,调出MS-DOS命令行,转到blast安装目录,输入命令“ blastn -version"即可查看版本。
2.blast本地数据库的构建
1.数据的获取
- 直接从NCBI或者其他数据库网站下载所需序列做成数据库,或者自己已有的测序数据(格式必须是fasta,名字可以自己随便命名)。
- 从NCBI中的ftp库下载所需要的某一个库或几个库(其链接为ftp://ftp.ncbi.nlm.nih.gov/blast/db/FASTA/)其中nr.gz为非冗余的数据库,nt.gz为核酸数据库,month.nt.gz为最近一个月的核酸序列数据。
- 利用新版blast自带的update_blastdb.pl进行下载,这需要安装perl程序。
上述三种方法各有优缺点:前两种下载速度较快,但是检索前都需要对数据库进行格式化(转化成二进制数据),第三种方法下载速度较慢,但是是NCBI中已经格式化好的,在进行本地检索时不需再进行格式化,直接用即可。
2.数据的格式化
以xk001.fasta作为查询序列,以nr.fasta作为数据库文件为例。首先将nr.fasta放到C:\Blast\db文件夹下,然后调出MS-DOS命令行,转到C:\Blast\db文件夹下运行格式化命令。
格式化nr.fasta命令:
C:\Blast\db>makeblastdb -in nr -dbtype prot -title "nr" -out NR
Building a new DB, current time: 08/28/2013 08:59:17
New DB name: T
New DB title: nr
Sequence type: Protein
Keep Linkouts: T
Keep MBits: T
Maximum file size: 1000000000B
Adding sequences from FASTA; added 32044604 sequences in 3134.69 seconds.
因此,本地数据库已经建立完毕。
3.blast的使用方法
以xk001.fasta作为查询序列,以nr.fasta作为数据库文件为例。
blsat运行命令:
C:\Blast>blastx.exe -db NR -query xk001.fasta –out xk001.out -evalue 0.00001 -max_target_seqs 5 -num_threads 4 -outfmt 11
注:将核酸序列比对到蛋白库需要用blastx,outfmt的格式选择11,11为ans.格式,此格式可以转化为其他格式文件。其他参数可以通过-h选择。
如果一切运行良好的话,待运行完毕,你将在blast文件下看到一个xk001.out的结果。
